Wenn du anfängst Bioinformatik zu studieren, wirst du über diese Themen hier auf jeden Fall mal stolpern.
Themen
Die Bioinformatik ist ein Gebiet mit vielen verschiedenen, sich teilweise überschneidenden Themen, von denen wir euch hier einige aufzählen.
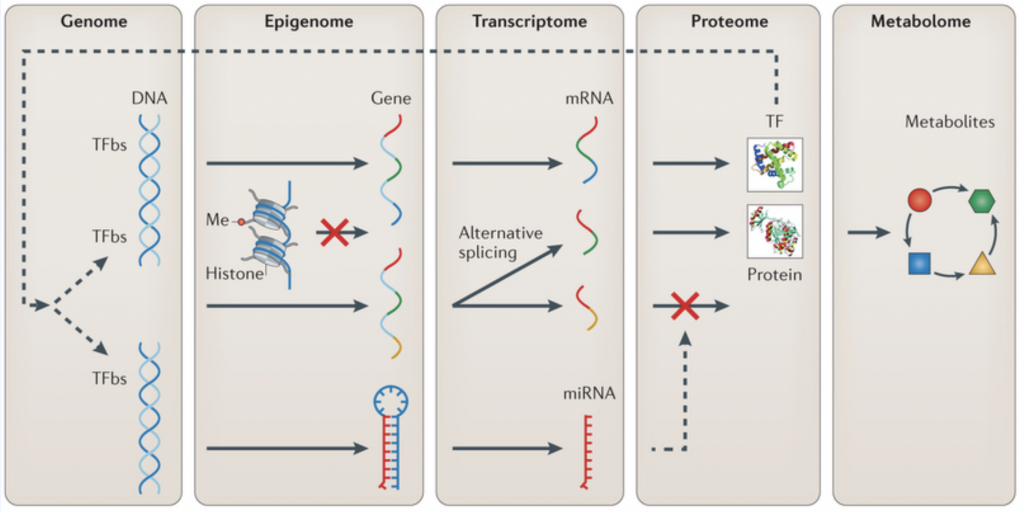
- Die „OMICS“ beschreiben im Groben die bioinformatischen Bereiche
- Genomics: Analyse von DNA-Sequenzen, Genen, genomische Variationen uvm.
- Transcriptomics: Analyse von RNA-Sequenzen, Genexpression, Transkriptvarianten, uvm.
- Proteomics: Analyse von Proteinsequenzen, Vorhersage von Proteinstrukturen, Interaktionen von Proteinen, Proteomen, uvm.
- Epigenomics: Analyse der Veränderungen in der Genaktivität, die nicht durch Veränderungen in der DNA-Sequenz verursacht werden (DNA-Methylierung, Histondeacetylierung usw.)
- Metabolomics: Analyse von Massenspektrometrie- und NMR-Daten, um Stoffwechselwege zu rekonstruieren und metabolische Netzwerke zu verstehen
Algorithmen
Generell, wenn du Algorithmen interessant findest und dir eine eigene Challenge setzen möchtest, kannst du auch das Skript zur „Algorithmischen Bioinformatik“ von Professor Heun durchlesen.
String Matching Algorithmen/“Pattern Finding“, um einzelne Strings („Zeichenketten“) in einem
Text zu finden
- Knuth-Morris-Pratt (KMP)
- Boyer-Moore (BM)

Sequenz Alignment Algorithmen, um 2 (oder mehr -> Multiples Sequenzen Alignment) unterschiedliche Sequenzen mit möglichst wenig falschen Buchstaben aneinander zu stellen (oder auch „alignen“). Damit können wir beispielsweise Basensequenzen von 2 Genen miteinander vergleichen. Beispielhafte Algorithmen sind:
- Needleman Wunsch: globales Alignment von zwei Sequenzen
- Smith Waterman: lokales Alignment von zwei Sequenzen
- BLAST: paarweises Alignment von multiplen Sequenzen zur Identifizierung von lokalen Ähnlichkeiten
- Hirschberg: Multiples Sequenzen Alignment unter Verwendung des Hirschberg-Algorithmus für globales Alignment
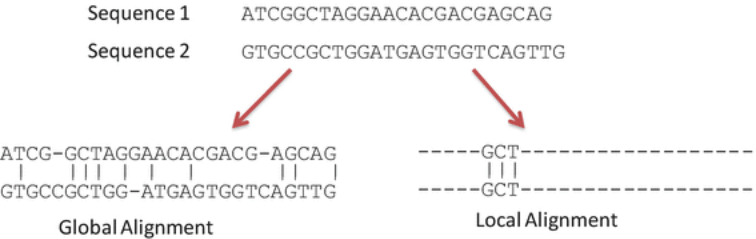
Kurz der Unterschied zwischen lokalem und globalem Alignment:
- globales Alignment: man versucht die ganze Länge der Sequenzen aneinander zu alignen
- lokales Alignment: man versucht einen Substring von einer Sequenz an eine andere zu alignen
Oft benutzt man dabei eine Kostenfunktion und versucht das möglichst kostengünstigste Ergebnis herauszufinden.
Beispiel:
Wir sagen, dass falsche Buchstaben aneinander zu stellen 2 kostet, eine Lücke kostet 1 und 2 richtige Charaktere aneinander zu stellen kostet nichts.
Wenn wir BAUM und RAUM miteinander alignen wollen, schauen wir uns erst die erste Stelle an (also B beim 1. Wort und R beim 2.).
Wenn wir die Buchstaben einfach so stehen lassen, kostet das 2. Wenn wir eine Lücke da hinstellen, kostet das 1. Dann müssen wir aber in beiden Wörtern eine Lücke hinzufügen, da sonst die Zeichenanzahl nicht mehr übereinstimmt. Das könnte so aussehen: